„Mimiviridae“ – Versionsunterschied
| [gesichtete Version] | [gesichtete Version] |
Ernsts (Diskussion | Beiträge) →Systematik: Eintrag gemäß MSL#38 und ICTV-Proposal zur Reorg der Imitervirales, Referenzen folgen, Merger danach... |
Ernsts (Diskussion | Beiträge) Info über extended Mimiviridae, d. h. externe Imitervirales in diesen neuen Artikel verschoben |
||
| Zeile 41: | Zeile 41: | ||
Für diese unmittelbare Verwandtschaft dieser herkömmlichen ''Mimiviridae'' wurde die informelle Bezeichnung „{{lang|en|Extended Mimiviridae}}“ ({{deS}} etwa „Erweiterte Mimiviridae“, auch ''Mimiviridae'' im weiteren Sinn [[sensu lato]] oder, veraltet, ''Megaviridae'') geschaffen.<ref name="Yutin2013" /> Diese Bezeichnung war ursprünglich vorgeschlagen worden, um insbesondere die Kandidaten einer „OLPG“ („{{lang|en|Organic Lake Phycodna Group}}“) genannten Klade aufzunehmen: Deren Vertreter waren ursprünglich den ''[[Phycodnaviridae]]'' in der (zwischenzeitlich geschaffenen) Ordnung ''Algavirales'', bis sich herausstellte, dass diese den ''Mimiviridae'' näherstehen als den ''Phycodnaviridae''. Dabei sind beide Gruppen, die ''Phycodnaviridae'' genauso wie die ''Mimiviridae'', Mitglieder der Klasse ''Megaviricetes''. |
Für diese unmittelbare Verwandtschaft dieser herkömmlichen ''Mimiviridae'' wurde die informelle Bezeichnung „{{lang|en|Extended Mimiviridae}}“ ({{deS}} etwa „Erweiterte Mimiviridae“, auch ''Mimiviridae'' im weiteren Sinn [[sensu lato]] oder, veraltet, ''Megaviridae'') geschaffen.<ref name="Yutin2013" /> Diese Bezeichnung war ursprünglich vorgeschlagen worden, um insbesondere die Kandidaten einer „OLPG“ („{{lang|en|Organic Lake Phycodna Group}}“) genannten Klade aufzunehmen: Deren Vertreter waren ursprünglich den ''[[Phycodnaviridae]]'' in der (zwischenzeitlich geschaffenen) Ordnung ''Algavirales'', bis sich herausstellte, dass diese den ''Mimiviridae'' näherstehen als den ''Phycodnaviridae''. Dabei sind beide Gruppen, die ''Phycodnaviridae'' genauso wie die ''Mimiviridae'', Mitglieder der Klasse ''Megaviricetes''. |
||
== Beschreibung und Systematik == |
|||
== ''Imitervirales'' == |
|||
Im März 2020 hat das ICTV dem Vorschlag von Koonin et al. vom April 2020 entsprochen und neben der Klasse ''Megaviricetes'' auch die darin enthaltene Ordnung '''''Imitervirales''''' (mit der einzigen Familie ''Mimiviridae'') geschaffen, um einen taxonomischen Rahmen für solche ''Mimiviridae''-ähnlichen Vertreter bereitzustellen.<ref group="Anm.">Die Schwesterordnung ''[[Algavirales]]'' innerhalb der ''Megaviricetes'' hat eine ähnliche Stellung und (potenteielle) Funktion für die ''Phycodnaviridae'' und ihre unmittelbare Verwandtschaft.</ref> |
|||
Zuvor hatte bereits Guglielmini et al. (2018/2019) die Bezeichnung „''Megavirales''“ in diesem engen Sinn gebraucht.<ref name="Guglielmini2019" /> |
|||
Kandidaten, die den ''Mimiviridae'' näherstehen als den ''Phycodnaviridae'', dürfen damit der Vermutung nach den ''Imitervirales'' zugerechnet werden, weil in der taxonomischen Rangstufe darüber, der Klasse ''Megaviricetes'', die ''Phycodnaviridae'' auch enthalten sind.<ref group="Anm.">Der Gebrauch der vom ICTV geschaffenen neuen Ordnungsbezeichnung ''Imitervirales'' ist noch nicht sehr verbreitet. Es gibt derzeit vielleicht weder Vorschläge, diese mit den „{{lang|en|Extended ''Mimiviridae''}}“ gleichzusetzen, noch für Vertreter die in der einen, nicht aber in der anderen Gruppe anzusiedeln wären. Faktisch bezeichnen beide daher dasselbe. Eine solche Gleichsetzung kann hier aber – solange unbelegt – nicht vorausgesetzt werden.</ref> |
|||
Die genauen [[Phylogenetik|phylogenetischen]] Beziehungen der Kandidaten für die „{{lang|en|Extended Mimiviridae}}“ werden derzeit immer noch diskutiert. Wenn die als OLPG bezeichnete Klade eine Schwestergruppe zu den Cafeteriaviren darstellen sollte, dann wäre diese genauso Mitglied der ''Mimiviridae'' wie die Cafeteriaviren. Sollten die OLPG-Kandidaten aber basaler stehen als ''Mimivirus'' und ''Cafeteriavirus'', dann wären sie nur ''Mimiviridae''-ähnlich und stünden außerhalb der ''Mimivirdae'' in der Ordnung ''Imitervirales''. |
|||
Obwohl erst einige wenige Mitglieder dieser Familie (bzw. der vorgeschlagenen Ordnung) im Detail beschrieben sind, erscheint es als wahrscheinlich, dass es weitere Virusarten gibt, die in diese Verwandtschaftsgruppe ([[Taxon]]) gehören,<ref name="Ghedin2005" /><ref name="Monier2008" /> insbesondere könnten weitere Kandidaten aus Metagenomanalysen folgen. Viele Viren sind bereits isoliert aber noch nicht ausreichend charakterisiert und daher vom ICTV noch nicht bestätigt. |
|||
Da bislang möglicherweise noch kein [[Virusisolierung|Isolat]] oder [[Contig]] (Genomsequenz aus der [[Metagenomik]]) explizit der Klasse ''Imitervirales'' zugeordnet wurde, gibt es faktisch keinen Unterschied zu den „{{lang|en|Extended Mimiviridae}}“. |
|||
=== ''Mimiviridae'' === |
|||
Vom ICTV bestätigte Mitglieder und vorgeschlagene Kandidaten der ''Mimiviridae'' (im herkömmlichen, engen Sinne): |
Vom ICTV bestätigte Mitglieder und vorgeschlagene Kandidaten der ''Mimiviridae'' (im herkömmlichen, engen Sinne): |
||
* '''''Mimiviridae'' Gruppe I''': Das erste Mitglied der Familie ''Mimiviridae'', ''Acanthamoeba polyphaga mimivirus'' (APMV) ([[Gattung (Biologie)|Gattung]] ''[[Mimivirus]]'', Linie A) wurde bereits 2003 entdeckt.<ref name="Suzan-Monti2006" /> Weitere vorgeschlagene Mitglieder der Gattung ''Mimivirus'' sind:<ref name="CNRS2019" /><ref name="MGaia2014" /> |
* '''''Mimiviridae'' Gruppe I''': Das erste Mitglied der Familie ''Mimiviridae'', ''Acanthamoeba polyphaga mimivirus'' (APMV) ([[Gattung (Biologie)|Gattung]] ''[[Mimivirus]]'', Linie A) wurde bereits 2003 entdeckt.<ref name="Suzan-Monti2006" /> Weitere vorgeschlagene Mitglieder der Gattung ''Mimivirus'' sind:<ref name="CNRS2019" /><ref name="MGaia2014" /> |
||
| Zeile 85: | Zeile 73: | ||
'''ICTV''': Vom ICTV bestätigt sind innerhalb der ''Mimiviridae'' mit Stand 10. April 2021 nur die beiden Gattungen ''Mimivirus'' mit der Spezies ''Acanthamoeba polyphaga mimivirus'' (APMV) und ''Cafeteriavirus'' mit der Spezies ''Cafeteria-roenbergensis-Virus'' (CroV). |
'''ICTV''': Vom ICTV bestätigt sind innerhalb der ''Mimiviridae'' mit Stand 10. April 2021 nur die beiden Gattungen ''Mimivirus'' mit der Spezies ''Acanthamoeba polyphaga mimivirus'' (APMV) und ''Cafeteriavirus'' mit der Spezies ''Cafeteria-roenbergensis-Virus'' (CroV). |
||
=== {{Anker|OLPG}}„OLPG“ === |
|||
[[Datei:Viruses-09-00040-g002b.png|mini|Dünnschliffe von ''[[Prymnesium parvum]]'' 946/6 48 h nach Infektion mit „''Prymnesium parvum DNA-Virus BW1''“ (PpDNAV-BW1). C: [[Chloroplast]]; V: [[kontraktile Vakuole]]; M: [[Mitochondrium|Mitochondrien]], P: [[Pyrenoid]]<ref name="Wagstaff2017" />]] |
|||
[[Datei:Viruses-09-00040-g002c.png|mini|Freie [[Virion]]en von „''Prymnesium parvum DNA-Virus BW1''“ (PpDNAV-BW1) in Kultur 72 h nach der Infektion<ref name="Wagstaff2017" />]] |
|||
* Eine „OLPG“ („{{lang|en|[[Organic Lake]] Phycodna Group}}“) genannte Gruppe war zunächst – wie diese vorläufigen Name besagt – vorschlagsgemäß der Familie ''[[Phycodnaviridae]]'' zugeordnet worden. Grund war, dass sie (im Gegensatz zu den damals bekannten Vertretern und Kandidaten der ''Mimiviridae'') Pflanzen oder Grünalgen statt [[Amöbe]]n parasitieren. Spätere genauere Analysen ergaben aber, dass die OLPG-Mitglieder offenbar eher mit den ''Mimiviridae'' verwandt sind.<ref name="Claverie2018" /><ref name="Yutin2013" /><ref name="Reyes2016" /><ref name="Wagstaff2017">Ben A. Wagstaff, Iulia C. Vladu, J. Elaine Barclay, Declan C. Schroeder, Gill Malin, Robert A. Field: [https://www.mdpi.com/1999-4915/9/3/40/htm Isolation and Characterization of a Double Stranded DNA Megavirus Infecting the Toxin-Producing Haptophyte ''Prymnesium parvum''], in: Viruses 2017, Band 9, Nr. 3, Special Issue Marine Viruses 2016, 40; [[doi:10.3390/v9030040]]</ref> |
|||
: Benannt wurde die Gruppe nach „''[[Organic Lake Phycodnavirus]] 1''“ und „''2''“ (OLPV1 und OLPV2), gefunden im [[Organic Lake]], [[Antarktis]]. Als Mitglied der „''{{lang|en|Extended Mimiviridae}}''“ wurde diese Klade gelegentlich auch als ''Mimiviridae'' Gruppe III bezeichnet (die Klosneuviren wurden erst später gefunden).<ref name="WZhang2015" /><ref name="Gornik2019" /> |
|||
: OLPG umfasst die folgenden vorgeschlagenen Kandidaten: |
|||
:# „''[[Organic Lake phycodnavirus]] 1''“ und „''2''“ (OLPV1 und OLPV2) |
|||
:# „''[[Chrysochromulina-ericina-Virus|Chrysochromulina ericina virus]] 01''“ (CeV-01B, CeV01 oder CeV, alias „''[[Haptolina]] ericina virus''“, HeV)<ref name="CNRS2018" /><ref name="Barik2018" /><ref name="Blanc-Mathieu2021" /> |
|||
:# „''[[Haptolina]] ericina virus RF02''“ (HeV-RF02)<ref name="Johannessen2015" /> |
|||
:# „''[[Phaeocystis]] globosa virus 12''“, „''14''“, „''16''“ (PgV-12T, PgV-14T, PgV-16T)<ref name="Claverie2018" /><ref name="Barik2018" /><ref name="JAbra2018" /><ref name="CNRS2019" /><ref name="Santini2013" /><ref name="Li2019" /> |
|||
:# „''Phaeocystis pouchetii virus 01''“ (PpV)<ref name="Yutin2013" /> |
|||
:# „''[[Yellowstone Lake mimivirus]]''“ (alias „''Yellowstone lake giant virus''“, YLGV,<br />veraltet „''Yellowstone Lake phycodnavirus 4''“, YSLPV4)<ref name="YLPV">NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1586712 ''Yellowstone lake mimivirus''] (species) – kein Mitglied der Gattung ''Mimivirus'', sondern ein „Mimivirus-like virus“ – ist offenbar identisch mit „''Yellowstone lake giant virus''“, YLGV, und „''Yellowstone Lake Phycodnavirus 4''“, YSLPV4, zu unterscheiden von dem nicht verwandten, nicht weiter klassifizierten [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgiid=1534536 Yellowstone hot spring archaeal RNA virus] (species) und den „''Yellowstone Lake [[Virophage]]s''“ (YSLVs)</ref><ref name="WZhang2015" /><ref name="Barik2018" /> |
|||
:# „''[[Prymnesium kappa]] virus RF01''“ (PkV-RF01)<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1499987 Prymnesium kappa virus] (species)</ref><ref name="Gallot2017" /><ref name="Johannessen2015" /><ref name="CNRS2019" /><ref name="Li2019" /> |
|||
:# „''[[Prymnesium parvum]] DNA virus BW1''“ (PpDNAV-BW1)<ref name="Li2019" /><ref name="Wagstaff2018" /> |
|||
:# gvSAG AB-572-A11<ref name="Wilson2017" /> |
|||
HeV-RF02 war (mit einem [[Kapsid]]<nowiki />durchmesser von ca. 310 [[Meter#nm|nm]]) zum Zeitpunkt seiner Entdeckung 2015 das größte bekannte Algenvirus.<ref name="Johannessen2015" /> |
|||
{{Absatz}} |
|||
=== {{Anker|AaV/ChoanoV}}Aureococcusviren === |
|||
Im September 2019 berichteten David M. Needham, Alexandra Z. Worden und Kollegen über das neu entdeckte „''[[Choanovirus]]''“ (ChoanoV 1 und 2), das ein virales [[Rhodopsin]] besitzt<ref name="NeedhamWorden2019" /> und zusammen mit dem „''[[Aureococcus anophagefferens]] virus''“ (AaV, {{enS|brown tide virus}})<ref name="Moniruzzaman2014" /><ref name="Moniruzzaman2020">Mohammad Moniruzzaman, Alaina R. Weinheimer, Carolina A. Martinez-Gutierrez, Frank O. Aylward: [https://www.nature.com/articles/s41586-020-2924-2 Widespread endogenization of giant viruses shapes genomes of green algae], in: [[nature]] vom 18. November 2020, [[doi:10.1038/s41586-020-2924-2]], dazu:<br />Kendall Daniels: Lurking in genomic shadows: How giant viruses fuel the evolution of algae, [https://vtnews.vt.edu/articles/2020/11/fralinlifesci-giant-viruses-fuel-the-evolution-of-algae.email.html vtnews], [https://scitechdaily.com/lurking-in-genomic-shadows-how-giant-viruses-fuel-the-genetic-evolution-of-organisms/ SciTechDaily], Quelle: Virginia Tech, 18. November 2020</ref><ref name="CNRS2019" /><ref name="Barik2018" /> |
|||
neben der „OLPG“ eine neue [[Klade]] innerhalb der „{{lang|en|Extended Mimiviridae}}“ bildet.<ref name="NeedhamWorden2019" /><ref>Jan Osterkamp: [https://www.spektrum.de/news/groesstes-riesenvirus-mit-mysterioeser-zusatzausstattung/1675384 Größtes Riesenvirus mit mysteriöser Zusatzausstattung], auf: Spektrum.de vom 23. September 2019</ref> Die Klade mit AaV wird von Schulz et al. (2020) informell als {{lang|en|Aureococcusviruses|de=Aureococcusviren}} bezeichnet.<ref name="Schulz2020" /> |
|||
=== {{Anker|PoV/TetV}}Tetraselmisviren === |
|||
[[Datei:TetV-2018-04-12-21.09.04-300x300.png|mini|[[Transmissionselektronenmikroskop|TEM]]-Aufnahme mit einer infizierten ''[[Tetraselmis]]''-Zelle im Querschnitt. Der Pfeil zeigt auf ein TetV-[[Virion]]. Maßstab 1 [[Meter#Mikrometer|μm]]. Inset: Negativ eines einzelnen TetV-Virions. Maßstab hier 200 [[Meter#nm|nm]].<ref name="Schvarcz2018" /><ref group="Anm." name="CC40">Das Material wurde von dieser Quelle kopiert, die unter einer [https://creativecommons.org/licenses/by/4.0/ Creative Commons Attribution 4.0 International License] verfügbar ist.</ref> ]] |
|||
Eine weitere Klade der {{lang|en|Extended Mimiviridae}} bilden die beiden Kandidaten „''[[Pyramimonas]] orientalis virus 01''“ (PoV bzw. PoV01),<ref name="Deeg2018" /><ref name="Schvarcz2018" /> und „''[[Tetraselmis]] virus''“ ({{nowrap|TetV-1}}).<ref name="Deeg2018" /><ref name="Schvarcz2018" /><ref name="scinexx2018-05" /><ref name="sciencedaily2018" /><ref name="CNRS2019" /> Die Klade mit TetV wird von Schulz ''et al.'' (2020) informell als {{lang|en|Tetraselmisviruses|de=Tetraselmisviren}} bezeichnet.<ref name="Schulz2020" /> Zu dieser Klade gehört offenbar auch „''[[Prymnesium kappa]] virus RF02''“ (PkV-RF02).<ref name="Johannessen2015" /> |
|||
=== DSLLAV1 === |
|||
Der vorerst jüngste Kandidat der „{{lang|en|Extended Mimiviridae}}“ ist „{{lang|en|Dishui Lake large alga virus 1}}“ (DSLLAV1). |
|||
Fundort: Dishui Lake {{Coordinate|NS=30.897227|EW=121.935294|type=waterbody|region=CN-SH|text=DEC|name=Dishui Hu}}, ein künstlich angelegter See in der Modellstadt [[Nanhui New City]], [[Pudong]], Schanghai<!--, Endstation der [[Metro Shanghai#Linie 16|Linie 16 der Metro Shanghai]]-->. |
|||
Nach Xu et al. (2020) steht dieser Kandidat basal in der Klade der erweiterten ''Mimiviridae''.<ref name="Xu2020" /> |
|||
== {{Anker|Mesomimiviridae|Mesomimivirinae}}Systematik == |
|||
=== Systematik mit MSL #38 vom 8. April 2023 === |
=== Systematik mit MSL #38 vom 8. April 2023 === |
||
* Familie ''[[Mimiviridae]]'' |
* Familie ''[[Mimiviridae]]'' |
||
| Zeile 178: | Zeile 132: | ||
:::* Spezies ''Mimivirus lagoaense'' mit<br |
:::* Spezies ''Mimivirus lagoaense'' mit<br |
||
/>Acanthamoeba polyphaga mimivirus Kroon (abgespaltet von Spezies ApMV) |
/>Acanthamoeba polyphaga mimivirus Kroon (abgespaltet von Spezies ApMV) |
||
=== Alte Systematik === |
=== Alte Systematik === |
||
| Zeile 291: | Zeile 244: | ||
<ref name="CNRS2019"> |
<ref name="CNRS2019"> |
||
[https://www.igs.cnrs-mrs.fr/wp-content/uploads/2019/03/TableVirus_Mars_2019.pdf ''List of the main “giant” viruses known as of today'' (March 2019).] (PDF) Centre national de la recherche scientifique, Université Aix Marseille, März 2019. |
[https://www.igs.cnrs-mrs.fr/wp-content/uploads/2019/03/TableVirus_Mars_2019.pdf ''List of the main “giant” viruses known as of today'' (March 2019).] (PDF) Centre national de la recherche scientifique, Université Aix Marseille, März 2019. |
||
</ref> |
|||
<ref name="sciencedaily2018"> |
|||
[https://www.sciencedaily.com/releases/2018/05/180503101643.htm ''A new giant virus found in the waters of Oahu, Hawaii''.] ScienceDaily, 3. Mai 2018 |
|||
</ref> |
</ref> |
||
<ref name="scinexx2018-02"> |
<ref name="scinexx2018-02"> |
||
[http://www.scinexx.de/wissen-aktuell-22465-2018-02-28.html ''Riesenviren an der Grenze zum Leben''], auf: scinexx.de, 28. Februar 2018 |
[http://www.scinexx.de/wissen-aktuell-22465-2018-02-28.html ''Riesenviren an der Grenze zum Leben''], auf: scinexx.de, 28. Februar 2018 |
||
</ref> |
|||
<ref name="scinexx2018-05"> |
|||
[https://www.scinexx.de/news/biowissen/neues-riesenvirus-entdeckt/ Neues Riesenvirus entdeckt - Vor Hawaii aufgespürtes Virus ist bisher größter Zellparasit pflanzlicher Organismen], auf: scinexx vom 4. Mai 2018 |
|||
</ref> |
</ref> |
||
<ref name="JAbra2018"> |
<ref name="JAbra2018"> |
||
| Zeile 314: | Zeile 261: | ||
<ref name="Bajrai2019"> |
<ref name="Bajrai2019"> |
||
Leena Hussein Bajrai, Saïd Mougari, Julien Andreani, Emeline Baptiste, Jeremy Delerce, Didier Raoult, Esam Ibraheem Azhar, Bernard La Scola, Anthony Levasseur; Joanna L. Shisler (Hrsg.): [https://www.researchgate.net/publication/336403536_Isolation_of_Yasminevirus_the_First_Member_of_Klosneuvirinae_Isolated_in_Coculture_with_Vermamoeba_vermiformis_Demonstrates_an_Extended_Arsenal_of_Translational_Apparatus_Components Isolation of Yasminevirus, the First Member of Klosneuvirinae Isolated in Coculture with ''Vermamoeba vermiformis'', Demonstrates an Extended Arsenal of Translational Apparatus Components], in: JVirol 94(1), Herbst 2019, [[doi:10.1128/JVI.01534-19]] |
Leena Hussein Bajrai, Saïd Mougari, Julien Andreani, Emeline Baptiste, Jeremy Delerce, Didier Raoult, Esam Ibraheem Azhar, Bernard La Scola, Anthony Levasseur; Joanna L. Shisler (Hrsg.): [https://www.researchgate.net/publication/336403536_Isolation_of_Yasminevirus_the_First_Member_of_Klosneuvirinae_Isolated_in_Coculture_with_Vermamoeba_vermiformis_Demonstrates_an_Extended_Arsenal_of_Translational_Apparatus_Components Isolation of Yasminevirus, the First Member of Klosneuvirinae Isolated in Coculture with ''Vermamoeba vermiformis'', Demonstrates an Extended Arsenal of Translational Apparatus Components], in: JVirol 94(1), Herbst 2019, [[doi:10.1128/JVI.01534-19]] |
||
</ref> |
|||
<ref name="Barik2018"> |
|||
Sailen Barik: [https://www.sciencedirect.com/science/article/pii/S2001037018300436 ''A Family of Novel Cyclophilins, Conserved in the Mimivirus Genus of the Giant DNA Viruses''.] In: ''Computational and Structural Biotechnology Journal'', Band 16, Juli 2018, S. 231–236, [[doi:10.1016/j.csbj.2018.07.001]] |
|||
</ref> |
</ref> |
||
<ref name="Bekliz2016"> |
<ref name="Bekliz2016"> |
||
Meriem Bekliz, Philippe Colson, Bernard La Scola: [https://www.mdpi.com/1999-4915/8/11/317/htm The Expanding Family of Virophages], in: MDPI Viruses 8(11), Special Issue Viruses of Protozoa, 23. November 2016, 317; [[doi:10.3390/v8110317]] |
Meriem Bekliz, Philippe Colson, Bernard La Scola: [https://www.mdpi.com/1999-4915/8/11/317/htm The Expanding Family of Virophages], in: MDPI Viruses 8(11), Special Issue Viruses of Protozoa, 23. November 2016, 317; [[doi:10.3390/v8110317]] |
||
</ref> |
</ref> |
||
<ref name="Blanc-Mathieu2021">Romain Blanc-Mathieu, Håkon Dahle, Antje Hofgaard, David Brandt, Hiroki Ban, Jörn Kalinowski, Hiroyuki Ogata, Ruth-Anne Sandaa: {{Webarchiv|url=https://journals.asm.org/doi/abs/10.1128/jvi.02446-20 |wayback=20210721015349 |text=A Persistent Giant Algal Virus, with a Unique Morphology, Encodes an Unprecedented Number of Genes Involved in Energy Metabolism |archiv-bot=2022-12-16 07:20:33 InternetArchiveBot }}, in: ASM Journals: Journal of Virology, Band 95, Nr. 8, 25. März 2021, [[doi:10.1128/JVI.02446-20]].<br />PrePrint: [https://www.biorxiv.org/content/10.1101/2020.07.30.228163v4 bioRxiv], [[doi:10.1101/2020.07.30.228163]], [[Cold Spring Harbor Laboratory|CSH]], 13. Januar 2021 (freier Volltext)</ref> |
|||
<ref name="Boudjemaa2020"> |
<ref name="Boudjemaa2020"> |
||
Hadjer Boudjemaa, Julien Andreani, Idir Bitam, Bernard La Scola: [https://www.mdpi.com/1424-2818/12/6/215/html ''Diversity of Amoeba-Associated Giant Viruses Isolated in Algeria''.] In: ''Diversity'', Band 12, Nr. 6, 215, 29. Mai 2020, [[doi:10.3390/d12060215]]. |
Hadjer Boudjemaa, Julien Andreani, Idir Bitam, Bernard La Scola: [https://www.mdpi.com/1424-2818/12/6/215/html ''Diversity of Amoeba-Associated Giant Viruses Isolated in Algeria''.] In: ''Diversity'', Band 12, Nr. 6, 215, 29. Mai 2020, [[doi:10.3390/d12060215]]. |
||
| Zeile 363: | Zeile 306: | ||
<ref name="Gallot-L2017"> |
<ref name="Gallot-L2017"> |
||
Lucie Gallot-Lavallée, Guillaume Blanc: ''A Glimpse of Nucleo-Cytoplasmic Large DNA Virus Biodiversity through the Eukaryotic Genomics Window''. In: ''Viruses'', 2017 Jan; 9(1), S. 17. {{PMC|5294986}}, PMID 28117696 |
Lucie Gallot-Lavallée, Guillaume Blanc: ''A Glimpse of Nucleo-Cytoplasmic Large DNA Virus Biodiversity through the Eukaryotic Genomics Window''. In: ''Viruses'', 2017 Jan; 9(1), S. 17. {{PMC|5294986}}, PMID 28117696 |
||
</ref> |
|||
<ref name="Gallot2017">Lucie Gallot-Lavallee, Guillaume Blanc, Jean-Michel Claverie: {{Webarchiv|url=https://jvi.asm.org/content/jvi/early/2017/04/21/JVI.00230-17.full.pdf |wayback=20191018171357 |text=Comparative genomics of ''Chrysochromulina Ericina'' Virus (CeV) and other microalgae-infecting large DNA viruses highlight their intricate evolutionary relationship with the established Mimiviridae family |archiv-bot=2022-12-16 07:20:33 InternetArchiveBot }} (PDF) in: J. Virol., 26 April 2017, [[doi:10.1128/JVI.00230-17]]</ref> |
|||
<ref name="Ghedin2005"> |
|||
E. Ghedin, J. M. Claverie: ''Mimivirus relatives in the Sargasso sea.'' In: ''Virol J.'' 2, 2005, S. 62. |
|||
</ref> |
</ref> |
||
<ref name="Gornik2019"> |
<ref name="Gornik2019"> |
||
Sebastian G. Gornik, Ian Hu, Imen Lassadi, Ross F. Waller: [https://www.mdpi.com/2076-2607/7/8/245/htm ''The Biochemistry and Evolution of the Dinoflagellate Nucleus''.] In: ''Microorganisms'', 7(8), 28. Juni 2019, S. 245, [[doi:10.3390/microorganisms7080245]]. |
Sebastian G. Gornik, Ian Hu, Imen Lassadi, Ross F. Waller: [https://www.mdpi.com/2076-2607/7/8/245/htm ''The Biochemistry and Evolution of the Dinoflagellate Nucleus''.] In: ''Microorganisms'', 7(8), 28. Juni 2019, S. 245, [[doi:10.3390/microorganisms7080245]]. |
||
</ref> |
|||
<ref name="Guglielmini2019"> |
|||
Julien Guglielmini, Anthony C. Woo, Mart Krupovic, [[Patrick Forterre]], Morgan Gaia: [https://www.pnas.org/content/116/39/19585 ''Diversification of giant and large eukaryotic dsDNnA viruses predated the origin of modern eukaryotes''.] In: ''PNAS'', 116(39), 10./24. September 2019, S. 19585–19592, [[doi:10.1073/pnas.1912006116]], PMID 31506349, [https://www.pnas.org/content/pnas/116/39/19585/F2.large.jpg Fig. 2]. Dazu:<br />Julien Guglielmini, Anthony Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: ''Diversification of giant and large eukaryotic dsDNA viruses predated the origin of modern eukaryotes''. 29. Oktober 2018, {{BioRxiv|10.1101/455816v1}}, [[doi:10.1101/455816]] (Preprint). |
|||
</ref> |
</ref> |
||
<ref name="Hauröder2018"> |
<ref name="Hauröder2018"> |
||
| Zeile 385: | Zeile 321: | ||
<ref name="Levasseur2016"> |
<ref name="Levasseur2016"> |
||
Anthony Levasseur, Meriem Bekliz, Eric Chabrière, Pierre Pontarotti, Bernard La Scola, Didier Raoult: ''MIMIVIRE is a defence system in mimivirus that confers resistance to virophage.'' In: ''[[Nature]]'', 2016, [[doi:10.1038/nature17146]]. |
Anthony Levasseur, Meriem Bekliz, Eric Chabrière, Pierre Pontarotti, Bernard La Scola, Didier Raoult: ''MIMIVIRE is a defence system in mimivirus that confers resistance to virophage.'' In: ''[[Nature]]'', 2016, [[doi:10.1038/nature17146]]. |
||
</ref> |
|||
<ref name="Li2019"> |
|||
Yanze Li, Hisashi Endo, Yasuhiro Gotoh, Hiroyasu Watai, Nana Ogawa, Romain Blanc-Mathieu, Takashi Yoshida, Hiroyuki Ogata: ''The Earth Is Small for “Leviathans”: Long Distance Dispersal of Giant Viruses across Aquatic Environments''. In: ''Microbes Environ.'', 34(3), September 2019, S. 334–339, Epub 3. August 2019, [[doi:10.1264/jsme2.ME19037]], {{PMC|6759346}}, PMID 31378760; insbes. [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6759346/figure/f4-34_334/?report=objectonly Fig. 4]; wegen der Platzierung im Stammbaum und DORTIGER Referenz auf Santini (2013) meint Phaeocystis_globosa_virus hier PgV-16T. |
|||
</ref> |
</ref> |
||
<ref name="Lingenhöhl2018"> |
<ref name="Lingenhöhl2018"> |
||
| Zeile 397: | Zeile 330: | ||
<ref name="Maruyama2016"> |
<ref name="Maruyama2016"> |
||
Fumito Maruyama, Shoko Ueki: [https://www.frontiersin.org/articles/10.3389/fmicb.2016.01942/full ''Evolution and Phylogeny of Large DNA Viruses, ''Mimiviridae'' and ''Phycodnaviridae'' Including Newly Characterized ''Heterosigma akashiwo Virus.] In: ''Front. Microbiol.'', 30. November 2016, [[doi:10.3389/fmicb.2016.01942]], {{PMC|5127864}}, PMID 27965659 |
Fumito Maruyama, Shoko Ueki: [https://www.frontiersin.org/articles/10.3389/fmicb.2016.01942/full ''Evolution and Phylogeny of Large DNA Viruses, ''Mimiviridae'' and ''Phycodnaviridae'' Including Newly Characterized ''Heterosigma akashiwo Virus.] In: ''Front. Microbiol.'', 30. November 2016, [[doi:10.3389/fmicb.2016.01942]], {{PMC|5127864}}, PMID 27965659 |
||
</ref> |
|||
<ref name="Monier2008"> |
|||
A. Monier, J. M. Claverie, H. Ogata: ''Taxonomic distribution of large DNA viruses in the sea.'' In: ''Genome Biol.'' 9(7), 2008, S. R106. |
|||
</ref> |
|||
<ref name="Moniruzzaman2014"> |
|||
Mohammad Moniruzzaman, Gary R. LeCleir, Christopher M. Brown, Christopher J. Gobler, Kay D. Bidle, William H. Wilson, Steven W. Wilhelm: [https://www.sciencedirect.com/science/article/pii/S0042682214002992?via%3Dihub ''Genome of brown tide virus (AaV), the little giant of the Megaviridae, elucidates NCLDV genome expansion and host–virus coevolution''.] In: ''Virology'', Band 466–467, Oktober 2014, online 14. Juli 29134, S. 60–70, [[doi:10.1016/j.virol.2014.06.031]] |
|||
</ref> |
</ref> |
||
<ref name="NeedhamWorden2019"> |
<ref name="NeedhamWorden2019"> |
||
| Zeile 418: | Zeile 345: | ||
<ref name="Racaniello2017"> |
<ref name="Racaniello2017"> |
||
Vincent Racaniello, David Tuller, Gertrud U. Rey: [http://www.virology.ws/2017/12/28/bodo-saltans-virus-an-abundant-giant-aquatic-mimivirus/ ''Bodo saltans virus, an abundant giant aquatic Mimivirus''.] virology blog, 28. Dezember 2017 |
Vincent Racaniello, David Tuller, Gertrud U. Rey: [http://www.virology.ws/2017/12/28/bodo-saltans-virus-an-abundant-giant-aquatic-mimivirus/ ''Bodo saltans virus, an abundant giant aquatic Mimivirus''.] virology blog, 28. Dezember 2017 |
||
</ref> |
|||
<ref name="Reyes2016"> |
|||
Carolina Reyes, Kenneth Stedman: [https://www.researchgate.net/post/Are_Phaeocystis_globosa_viruses_OLPG_and_Organic_Lake_phycodnavirus_a_part_of_the_Phycodnaviridae_or_Mimiviridae ''Are Phaeocystis globosa viruses (OLPG) and Organic Lake phycodnavirus a part of the Phycodnaviridae or Mimiviridae?''] Blog auf ResearchGate, 8. Januar 2016 |
|||
</ref> |
</ref> |
||
<ref name="Rolland2019"> |
<ref name="Rolland2019"> |
||
Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That ''Modified the Perception of the Virosphere''. In: ''Viruses'', 11(4), März/April 2019, pii: E312, [[doi:10.3390/v11040312]], {{PMC|6520786}}, PMID 30935049. <small>Anm.: Die „''Klosneuvirinae''“ sind per Namensendung eine (vorgeschlagene) Unterfamilie der ''Mimiviridae'', erst recht können die vorgeschlagenen Vertreter ''[[Yasminevirus]]'' und ''[[Fadolivirus]]'' nicht auch selbst neue Familien repräsentieren. Sie sind nicht die ersten isolierten Vertreter dieser '''Unter'''familie, das ist – wie an anderer stelle korrekt festgestellt wird, ''[[Bodo-saltans-Virus]]''.</small> |
Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That ''Modified the Perception of the Virosphere''. In: ''Viruses'', 11(4), März/April 2019, pii: E312, [[doi:10.3390/v11040312]], {{PMC|6520786}}, PMID 30935049. <small>Anm.: Die „''Klosneuvirinae''“ sind per Namensendung eine (vorgeschlagene) Unterfamilie der ''Mimiviridae'', erst recht können die vorgeschlagenen Vertreter ''[[Yasminevirus]]'' und ''[[Fadolivirus]]'' nicht auch selbst neue Familien repräsentieren. Sie sind nicht die ersten isolierten Vertreter dieser '''Unter'''familie, das ist – wie an anderer stelle korrekt festgestellt wird, ''[[Bodo-saltans-Virus]]''.</small> |
||
</ref> |
|||
<ref name="Santini2013"> |
|||
Sebastien Santini, Sandra Jeudy, Julia Bartoli, Olivier Poirot, Magali Lescot, Chantal Abergel, Valérie Barbe, K. Eric Wommack, Anna A. M. Noordeloos, Corina P. D. Brussaard, Jean-Michel Claverie: ''Genome of Phaeocystis globosa virus PgV-16T highlights the common ancestry of the largest known DNA viruses infecting eukaryotes''. In: ''Proc Natl Acad Sci USA'', 110(26), 25. Juni 2013, S. 10800–10805, Epub 10. Juni 2013, [[doi:10.1073/pnas.1303251110]], {{PMC|3696832}}, PMID 23754393 |
|||
</ref> |
</ref> |
||
<ref name="Schulz2017"> |
<ref name="Schulz2017"> |
||
| Zeile 449: | Zeile 370: | ||
<ref name="Schulz2020"> |
<ref name="Schulz2020"> |
||
Frederik Schulz, Simon Roux, David Paez-Espino, Sean Jungbluth, David A. Walsh, Vincent J. Denef, Katherine D. McMahon, Konstantinos T. Konstantinidis, Emiley A. Eloe-Fadrosh, Nikos C. Kyrpides, Tanja Woyke: [https://www.nature.com/articles/s41586-020-1957-x ''Giant virus diversity and host interactions through global metagenomics''.] In: Nature Band 578, S. 432–436, 22. Januar 2020, [[doi:10.1038/s41586-020-1957-x]], insbes. [https://www.nature.com/articles/s41586-020-1957-x/figures/1 Fig. 1] |
Frederik Schulz, Simon Roux, David Paez-Espino, Sean Jungbluth, David A. Walsh, Vincent J. Denef, Katherine D. McMahon, Konstantinos T. Konstantinidis, Emiley A. Eloe-Fadrosh, Nikos C. Kyrpides, Tanja Woyke: [https://www.nature.com/articles/s41586-020-1957-x ''Giant virus diversity and host interactions through global metagenomics''.] In: Nature Band 578, S. 432–436, 22. Januar 2020, [[doi:10.1038/s41586-020-1957-x]], insbes. [https://www.nature.com/articles/s41586-020-1957-x/figures/1 Fig. 1] |
||
</ref> |
|||
<ref name="Schvarcz2018"> |
|||
Christopher R. Schvarcz, Grieg F. Steward: ''A giant virus infecting green algae encodes key fermentation genes''. In: ''Virology'', Band 518, Mai 2018, S. 423–433, [[doi:10.1016/j.virol.2018.03.010]].<br /> |
|||
Vincent Racaniello: [https://www.virology.ws/2018/04/12/fermentation-genes-in-a-giant-algal-virus/ ''Fermentation genes in a giant algal virus''.] virology blog, 12. April 2018. |
|||
</ref> |
</ref> |
||
<ref name="Suzan-Monti2006"> |
<ref name="Suzan-Monti2006"> |
||
M. Suzan-Monti, B. La Scola, D. Raoult: ''Genomic and evolutionary aspects of Mimivirus.'' In: ''Virus Res.'' 117(1), 2006, S. 145–155. |
M. Suzan-Monti, B. La Scola, D. Raoult: ''Genomic and evolutionary aspects of Mimivirus.'' In: ''Virus Res.'' 117(1), 2006, S. 145–155. |
||
</ref> |
|||
<ref name="Wagstaff2018"> |
|||
Ben A Wagstaff, Edward S. Hems, Martin Rejzek, Jennifer Pratscher, Elliot Brooks, Sakonwan Kuhaudomlarp, Ellis C. O’Neill, Matthew I. Donaldson, Steven Lane, John Currie, Andrew M Hindes, Gill Malin, J. Colin Murrell, Robert A. Field: ''Insights into toxic Prymnesium parvum blooms: the role of sugars and algal viruses''. In: ''Biochem Soc Trans'', 46(2), 17. April 2018, S. 413–421, [[doi:10.1042/BST20170393]], Epub 14. März 2018, PMID 29540506, {{PMC|5906706}} |
|||
</ref> |
</ref> |
||
<ref name="Wilson2017"> |
<ref name="Wilson2017"> |
||
Version vom 30. April 2023, 19:12 Uhr
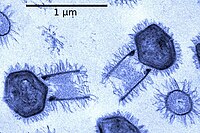
| Mimiviridae | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Mimiviridae | ||||||||||||||||
| Links | ||||||||||||||||
|


Mimiviridae ist eine Familie von Riesenviren der Klasse Megaviricetes im Phylum Nucleocytoviricota (ursprünglich vorgeschlagen als „Nucleocytoplasmaviricota“), mit dem das International Committee on Taxonomy of Viruses (ICTV) die zuvor informell als Nucleocytoplasmic large DNA viruses (NCLDVs) bezeichnete Klade (Verwandtschaftsgruppe) offiziell in die Taxonomie der Viren aufgenommen hat.[1][Anm. 1] Wie bei allen NCLDV-Riesenviren ist das Genom der „Extended Mimiviridae“ unsegmentiert (monopartit) und besteht aus einem linearen Doppelstrang-DNA-Molekül mit großer Länge (wie für Riesenviren üblich).
Zur Familie gehören derzeit (Stand 12. April 2021) vom ICTV bestätigt nur die beiden Gattungen Mimivirus und Cafeteriavirus mit jeweils einer Spezies (Art), Acanthamoeba-polyphaga Mimivirus bzw. Cafeteria-roenbergensis Virus an. Es gibt aber eine große Zahl weiterer vorgeschlagener Kandidaten, die entweder direkt zur Familie Mimiviridae oder in deren unmittelbare Verwandtschaft gehören. Die Familie Mimiviridae (im herkömmlichen engen Sinne, sensu stricto) ist dabei zu verstehen aks die kleinste Klade, die die ICTV-bestätigten Gattungen Mimivirus und Cafeteriavirus umfasst.
Für diese unmittelbare Verwandtschaft dieser herkömmlichen Mimiviridae wurde die informelle Bezeichnung „Extended Mimiviridae“ (deutsch etwa „Erweiterte Mimiviridae“, auch Mimiviridae im weiteren Sinn sensu lato oder, veraltet, Megaviridae) geschaffen.[3] Diese Bezeichnung war ursprünglich vorgeschlagen worden, um insbesondere die Kandidaten einer „OLPG“ („Organic Lake Phycodna Group“) genannten Klade aufzunehmen: Deren Vertreter waren ursprünglich den Phycodnaviridae in der (zwischenzeitlich geschaffenen) Ordnung Algavirales, bis sich herausstellte, dass diese den Mimiviridae näherstehen als den Phycodnaviridae. Dabei sind beide Gruppen, die Phycodnaviridae genauso wie die Mimiviridae, Mitglieder der Klasse Megaviricetes.
Beschreibung und Systematik
Vom ICTV bestätigte Mitglieder und vorgeschlagene Kandidaten der Mimiviridae (im herkömmlichen, engen Sinne):
- Mimiviridae Gruppe I: Das erste Mitglied der Familie Mimiviridae, Acanthamoeba polyphaga mimivirus (APMV) (Gattung Mimivirus, Linie A) wurde bereits 2003 entdeckt.[4] Weitere vorgeschlagene Mitglieder der Gattung Mimivirus sind:[5][6]
- Spezies „Mamavirus“ (ACMV), „Niemeyer-Virus“ (NYMV), „Hirudovirus“, „Fauteuil-Virus“, „Lactours-Virus“, „Longchamps-Virus“, „Terravirus 2“ (ebenfalls zur Gattung Mimivirus Linie A),
- Spezies „Moumouvirus“ (Gattung Mimivirus Linie B)
- Spezies „Megavirus chilensis“ mit Subtypen Courdovirus 5, 7, 11, und Terravirus 1 (Gattung Mimivirus Linie C)
- In die Verwandtschaft der Gattung Mimivirus gehören per Vorschlag auch die später entdeckten Kandidaten „Tupanvirus“[7][8][9][10][11] und Platanovirus. Nach verschiedenen neueren Arbeiten steht diese Tupanvirus-Gruppe jedoch der Gattung Mimivirus als Schwestergruppe gegenüber.[12][8]
- Zusammen bilden alle diese Vertreter sie die Klade Mimiviridae Gruppe I, für die vorgeschlagen wurde, ihr den Rang einer Unterfamilie mit der Bezeichnung „Megamimivirinae“[7] oder „Mimivirinae“[13] zu vergeben. Vom ICTV offiziell bestätigt sind von dieser Gruppe bislang die Gattung Mimivirus mit der Spezies Acanthamoeba polyphaga mimivirus (APMV).
- Mimiviridae Gruppe II: Ebenfalls vom ICTV bestätigt sind die Gattung Cafeteriavirus mit der Spezies Cafeteria-roenbergensis-Virus (CroV). Zusammen mit einigen Vorschlägen in dieser Gattung oder in der unmittelbaren Verwandtschaft bilden sie die Klade Mimiviridae Gruppe II, für die ebenfalls der Rang einer (noch unbenannten) Unterfamilie vorgeschlagen wurde.[14]
- Im Februar 2013 schlugen S. Clouthier et al. für das von ihnen gefunden „Namao-Virus“ (NV), das Störe (englisch sturgeos) wie den See-Stör (Acipenser fulvescens) parasitiert, eine Gruppe vor mit der provisorischen Bezeichnung „sturgeon Nucleocytoplasmic Large DNA Virus“ (sNCLDV). NV konnte innerhalb der Mimiviridae keiner der bekannten Gruppen zugeordnet werden. Phylogenetische Analysen zeigten aber, dass NV und das Cafeteria-roenbergensis-Virus (CroV) Schwestertaxa sein sollten.[15][16][17][18] Die Klade mit NV wird von Schulz et al. (2020) informell als Namaoviruses ‚Namaoviren‘ bezeichnet.[19]
- Klosneuviren: Eine weitere Klade der Mimiviridae sind die 2017 zunächst nur durch Metagenomik entdeckten Klosneuviren mit den Kandidaten „Klosneuvirus“, „Catovirus“, „Indivirus“ und „Hokovirus“.[20] Manche Autoren schlagen vor, die Klade der Klosneuviren taxonomisch als Unterfamilie Klosneuvirinae einzustufen.[21][22]
Später hinzugekommene Kandidaten sind Bodo-saltans-Virus (BsV),[23] Fadolivirus[24] und Yasminevirus[25] (aus Isolaten unter Zuhilfenahme von Wirtszellkulturen)[22]
Ebenfalls in die Verwandtschaft der Klosneuviren passen zwei Contigs aus Metagenomanalysen des Schwarzen Rauchers „Lokis Schloss“ (englisch Loki’s Castle),[Anm. 2] LCMiAC01 und LCMiAC02.[26]
- Nach der Sichtweise der meisten Autoren werden die Klosneuviren als Unterfamilie mit der vorläufigen Bezeichnung „Klosneuvirinae“ eingestuft.[21][22]
- „gvSAG AB-566-O17“: Der von Wilson et al. 2017 beschriebene Kandidat „gvSAG AB-566-O17“ (vom NCBI als nicht näher klassifizierte Mimiviridae-Spezies mit Mimivirus AB-566-O17 bezeichnet[27]) gehört nicht der Gattung Mimivirus an, sondern ist als Kandidat der Mimiviridae ein „Mimivirus-like virus“. Nach den Autoren (Fig. 2) ist es mit APMV weitläufiger verwandt als CroV, aber näher als PgV-16T und AaV. Es ist daher keiner dieser Gruppen zuzuordnen, sondern steht entweder basal in den Mimivirdae s. s. oder bildet ein Schwestertaxon zu diesen.[28]
- Mimiviridae aus Waldbodenproben: Im November 2018 berichteten Frederik Schulz und Kollegen über die Entdeckung von 16 neuen Riesenviren per Metagenomanalyse von Waldbodenproben. Für diese Viren wurden vorläufige Namen vergeben, die meist auf ihre Herkunft hinweisen. Zur Familie der Mimiviridae scheinen davon die folgenden zu gehören:[7][13][29]
- Gruppe I: „Satyrvirus“ (Tupanvirus-Gruppe),[30]
- Gruppe II: „Faunusvirus sp.“ (nicht zu verwechseln mit der offiziellen Gattung Faunusvirus der Chaseviridae),[31]
- Klosneuviren: „Gaeavirus“,[32] „Homavirus“,[33] „Barrevirus“,[34] „Dasosvirus“,[35] „Edafosvirus“,[36] „Terrestrivirus“,[37] „Harvfovirus“[38] und „Hyperionvirus“.[39]
Diskussion: Manche Autoren sehen eine enge Verwandtschaft der Klosneuviren mit den Cafeteriaviren (inkl. „Namao-Virus“) und schlagen daher eine gemeinsame Unterfamilie „Aquavirinae“ vor.[40][5]
Deeg et al. (2018) stufen die herkömmlichen Mimiviridae herab zu einer Unterfamilie „Megavirinae“, in der die Klosneuviren und die Mimiviridae Gruppe I Schwesterkladen ohne Rang sind. In der Familie Mimiviridae ist dann noch Platz für weitere Mitglieder und Unterfamilien (s. u.).[13]
ICTV: Vom ICTV bestätigt sind innerhalb der Mimiviridae mit Stand 10. April 2021 nur die beiden Gattungen Mimivirus mit der Spezies Acanthamoeba polyphaga mimivirus (APMV) und Cafeteriavirus mit der Spezies Cafeteria-roenbergensis-Virus (CroV).
Systematik mit MSL #38 vom 8. April 2023
- Familie Mimiviridae
- Unterfamilie Aliimimivirinae (Mimiviridae Gruppe II, Cafeteriaviren)
- Gattung Rheavirus (früher Cafeteriavirus)
- Spezies Rheavirus sinusmexicani (Cafeteria-roenbergensis-Virus, CroV)
mit Cafeteria roenbergensis virus BV-PW1
- Spezies Rheavirus sinusmexicani (Cafeteria-roenbergensis-Virus, CroV)
- Gattung Rheavirus (früher Cafeteriavirus)
- Unterfamilie Klosneuvirinae (Klosneuviren, engl. Klosneuviruses)[20]
- Gattung Fadolivirus
- Spezies Fadolivirus algeromassiliense
mit Fadolivirus FV1/VV64
- Spezies Fadolivirus algeromassiliense
- Gattung Theiavirus
- Spezies Theiavirus salishense (Bodo-saltans-Virus, BsV)
mit Bodo saltans virus strain NG1
- Spezies Theiavirus salishense (Bodo-saltans-Virus, BsV)
- Gattung Yasminevirus
- Spezies Yasminevirus saudimassiliense
mit Yasminevirus sp. GU-2018
- Spezies Yasminevirus saudimassiliense
- Gattung Fadolivirus
- Unterfamilie Megamimivirinae (zu Mimiviridae Gruppe I, Mimiviren s. l.)
- Gattung Cotonvirus
- Spezies Cotonvirus japonicum
mit Cotonvirus japonicusT
- Spezies Cotonvirus japonicum
- Gattung Megavirus (Mimiviridae Gruppe I, Linie C)
- Spezies Megavirus baoshanense mit
Megavirus baoshan strain SH - Spezies Megavirus chilense mit
Megavirus chilensisT, Megavirus lba isolate LBA111,
Megavirus terra1, Megavirus courdo11,
Megavirus vitis isolate vigne,
„Acanthamoeba polyphaga mimivirus“ ASM381511v1 (verschoben),
„Acanthamoeba polyphaga mimivirus“ ASM381513v1 (verschoben)
- Spezies Megavirus baoshanense mit
- Gattung Cotonvirus
- Unterfamilie Aliimimivirinae (Mimiviridae Gruppe II, Cafeteriaviren)
- Spezies Megavirus powaiense (Powai lake megavirus, PLMV) mit
Powai lake megavirus isolate 1
- Spezies Megavirus powaiense (Powai lake megavirus, PLMV) mit
- Gattung Moumouvirus (Mimiviridae Gruppe I, Linie B)
- Spezies Moumouvirus australiense mit
Moumouvirus australiensis isolate 10A - Spezies Moumouvirus goulettemassiliense mit
Moumouvirus goulette - Spezies Moumouvirus moumou mit
Acanthamoeba polyphaga moumouvirusT, Saudi moumouvirus
- Spezies Moumouvirus australiense mit
- Gattung Tupanvirus (Mimiviridae Gruppe I, Tupanvirus-Linie, Tupanviren)
- Spezies Tupanvirus altamarinense mit
Tupanvirus deep ocean - Spezies Tupanvirus salinum mit
Tupanvirus soda lake
- Spezies Tupanvirus altamarinense mit
- Unterfamilie nicht zugewiesen (zu Mimiviridae Gruppe I, Mimiviren s. l.)
- Gattung Mimivirus (Mimiviridae Gruppe I, Linie A)
- Spezies Mimivirus bradfordmassiliense (Acanthamoeba-polyphaga-Mimivirus, en. Acanthamoeba polyphaga mimivirus, ApMV) mit
Acanthamoeba polyphaga mimivirusT,
Acanthamoeba polyphaga mimivirus M4,
Acanthamoeba polyphaga mimivirus Oyster,
Acanthamoeba polyphaga mimivirus strain Amazonia,
Acanthamoeba polyphaga mimivirus ViralProj60053,
Acanthamoeba castellanii mamavirus Hal-V,
Acanthamoeba castellanii mimivirus kasaii,
Acanthamoeba castellanii mimivirus shirakomae,
Mimivirus terra2, Mimivirus Bombay isolate 1,
Hirudovirus strain Sangsue, Niemeyer virus (NYMV),
Samba virus
- Spezies Mimivirus bradfordmassiliense (Acanthamoeba-polyphaga-Mimivirus, en. Acanthamoeba polyphaga mimivirus, ApMV) mit
- Gattung Mimivirus (Mimiviridae Gruppe I, Linie A)
- Spezies Mimivirus lagoaense mit
Acanthamoeba polyphaga mimivirus Kroon (abgespaltet von Spezies ApMV)
- Spezies Mimivirus lagoaense mit
Alte Systematik
Die genauen phylogenetischen Beziehungen der oben genannten Kladen und Kandidaten blieben lange Zeit unklar.
In den meisten jüngeren Veröffentlichungen wird jedoch angenommen, dass die genannten Kandidaten bis auf das basale DSLLAV1 zusammen eine monophyletische Gruppe bilden. Für diese war mit dem Rang einer Unterfamilie innerhalb einer Familie Megaviridae (aufgefasst als erweiterte Familie Mimiviridae) die Bezeichnung „Mesomimivirinae“ vorgeschlagen worden.[40][5][41] Beispielsweise sind bei Deeg et al. (2018) mit den zur Unterfamilie „Megavirinae“ herabgestuften herkömmlichen Mimiviridae dann die „Mesomimivirinae“ deren Schwester-Unterfamilie (innerhalb einer in sehr weitem Sinn verstandenen Familie Mimiviridae).[13]
Manche Autoren benutzen die informelle Bezeichnung Mimiviridae Gruppe III im Gebrauch, entweder im engen Sinn als neue, bessere Bezeichnung für den nicht mehr zutreffenden Terminus OLPG,[42][43] oder im weiteren Sinn als informelle Bezeichnung für die gesamte (monophyletisch verstandene) Klade „Mesomimivirinae“, wenn kein taxonomischer Rang für diese festgelegt werden soll.[44]
Nach der Einrichtung der höheren Rangstufen durch das ICTV dürfte die Entwicklung aber unter Berücksichtigung der hohen Diversität in die andere Richtung gehen: Die Mimiviridae werden in diesem Szenario weiter im herkömmlichen, engen Sinn verstanden. Für die neue Klade wird dann der Rang einer Familie Mesomimiviridae vorgeschlagen,[45] in diesem Szenario eine Schwesterfamilie der Mimiviridae (innerhalb der Ordnung Imitervirales).[40][8][46][7] Basal zu beiden Familien stünde dann DSLLAV1.[44]
Die folgende innere Systematik beruht im Wesentlichen auf dem Vorschlag von F. Schulz et al. (2018)[7] und (2020)[19] zur Ergänzung der ICTV-Nomenklatur.[47] Die von diesem aus der Metagenomanalyse von Waldboden stammenden Kandidaten sind grün wiedergegeben.
Die Tupanvirus-Gruppe in Übereinstimmung mit verschiedenen neueren Arbeiten basal in den Megamimivirinae dargestellt.[12][8]
| Imitervirales: Extended Mimiviridae |
| ||||||||||||||||||||||||||||||||||||||||||||||||

Clara Rolland et al. (2019) haben ein etwas abweichende Kladogramm veröffentlicht. Der Hauptunterschied zur obigen Version besteht darin, dass die Cafeteriaviren zur Mimiviridae-Erweiterung der „OLPG“ verschoben sind und TetV basal in den erweiterten Mimviridae (bzw. Imitervirales) steht. Faunusvirus sp. steht zudem basal in den Die in dieser Arbeit vorgestellten neuen Kandidaten „Fadolivirus“ und „Yasminevirus“ sind oben gemäß Vorschlag der Autoren den „Klosneuvirinae“ zugeordnet.[22]
Nach Claviere et al. (2018) ist Namao-Virus u. U. nicht Mitglied der Gruppe der Cafeteriaviren, sondern bildet eine Schwestergruppe derselben, der Klosneuvirinae (dort als new subfamily bezeichnet), der Megavirinae (bei basal stehenden Mesomimiviridae, Aureococcus anophagefferens virus und Tetraselmis virus).[18] Möglicherweise ist auch die Gattung Raphidovirus (zumindest der Vertreter Heterosigma akashiwo virus strain HaV53) nicht bei den Phycodnaviridae, sondern in einer eigenen Gruppe, eventuell zusammen mit Aureococcus anophagefferens virus anzusiedeln.[50][51] Für den phylogenetischen Baum der Mesomimiviridae kommen die Autoren basierend auf MutS7 (Fig. 2) zu einem vergleichbaren Ergebnis wie oben, basierend auf DNA-Polymerase B (Fig 1) gibt es jedoch Abweichungen: Hier stehen TetV und danach AaV basal in den Mimiviridae, die Mesomimiviridae als Verwandtschaftsgruppe reduzieren sich daher auf die „OLPG“.
Nach Johannessen et al. (2015) ist Prymnesium kappa virus RF01 (PkV-RF01, bisher Prymnesiovirus) eher ein Schwestertaxon von Pyramimonas orientalis virus 01 (PoV01), beide ohne Zuordnung zu den Phycodnaviridae oder den erweiterten Mimiviridae. Prymnesium kappa virus RF02 (PkV-RF02, bisher ebenfalls Prymnesiovirus) ist dagegen als Schwestertaxon von Phaeocystis globosa virus (mutmaßlich PgV-16T gemeint) ein Mitglied der „OLPG“ und damit der „Mesomimiviridae“.[52]
Nach Gallot-Lavallée1 und Blanc 2017 stehen die „OLPG“ basal in der hier „Mesomimiviridae“ bezeichneten Erweiterung der „Mimiviridae“, erst danach erfolgt die Gabelung der beiden Gruppen mit AaV bzw. PoV.[53]
Merkmale der Mimiviridae im Vergleich
| Virus | Aminoacyl-tRNA-Synthetase | Octocorallia-ähnliche MutS | Proteinfilamente (Länge) | Stargate[54] | Bekannter Virophage[55] | Cytoplasmische Virion-Fabrik |
Wirt |
|---|---|---|---|---|---|---|---|
| Megavirus chilensis (MVc) | 7 (Tyr, Arg, Met, Cys, Trp, Asn, Ile) | ja | ja (75 nm) | ja | nein | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Mamavirus AcMV | 4 (Tyr, Arg, Met, Cys) | ja | ja (120 nm) | ja | ja | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Mimivirus ApMV (Wildtyp M1) |
4 (Tyr, Arg, Met, Cys) | ja | ja (120 nm) | ja | ja | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Mimivirus M4 (bald/fiberless Variante) |
2 (Met, Cys) | nein | nein | ja | resistent | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Cafeteria-roenbergensis-Virus (CroV) | 1 (Ile) | ja | nein | nein | ja | ja | Phagotrophische Protozoen (Heterokonta, Stramenopiles) |
Virophagen
Es gibt Satellitenviren, die den Syntheseapparat der Mimiviridae für ihre eigene Vermehrung nutzen und Virophagen genannt werden, wenn sie ihren Helferviren (Wirtsviren) schaden.
- Der zuerst entdeckte Fall dieser Art siehe ist das Sputnikvirus. Dieses vermehrt sich natürlich nicht im Virion des Mimivirus, sondern nutzt den durch das Mimivirus umgestalteten Proteinsyntheseapparat der Wirtszelle (das Viroplasma) und ist von den Replikationsenzymen des Mimivirus abhängig.
- Ein weiterer Virophage namens „Zamilon“ befällt einen anderen „Mamavirus“ getauften Vertreter der Mimiviridae.[6] Inzwischen ist bekannt, dass Mimiviren der Gruppen I B und I C von Zamilon befallen werden, Mimiviren der Gruppe I A jedoch eine Resistenz (genannt MIMIVIRE, englisch mimivirus virophage resistance element) aufweisen, die ähnlich funktioniert wie das CRISPR/Cas-System.[56][57]
- „Sissivirophage“ dürfte seinem Namen entsprechend „Sissivirus“ infizieren.[22]
Die Virophagen der Riesenviren sind im Vergleich zu Satellitenviren anderer Helferviren ebenfalls vergleichsweise riesig und haben auch ein komplexeres Genom.[41][58]
Weblinks
- Christelle Desnues, Bernard La Scola, Natalya Yutin, Ghislain Fournous, Catherine Robert, Saïd Azza, Priscilla Jardot, Sonia Monteil, Angélique Campocasso, Eugene V. Koonin, and Didier Raoult: Provirophages and transpovirons as the diverse mobilome of giant viruses, in: PNAS 109 (44) vom 30. Oktober 2012, S. 18078–18083, doi:10.1073/pnas.1208835109
- Ana Cláudia dos S. P. Andrade, Thalita S. Arantes, Rodrigo A. L. Rodrigues, Talita B. Machado, Fábio P. Dornas, Melissa F. Landell, Cinthia Furst, Luiz G. A. Borges, Lara A. L. Dutra, Gabriel Almeida, Giliane de S. Trindade, Ivan Bergier, Walter Abrahão, Iara A. Borges, Juliana R. Cortines, Danilo B. de Oliveira, Erna G. Kroon, Jônatas S. Abrahão: Ubiquitous giants: a plethora of giant viruses found in Brazil and Antarctica, in: Virology Journal, Band 15, Nr. 22, 24. Januar 2018, doi:10.1186/s12985-018-0930-x
Anmerkungen
- ↑ Die Supergruppe der NCLDVs war zunächst als Ordnung „Megavirales“ (s. l.) vorgeschlagen worden – das war noch vor der Einrichtung höherer taxonomischer Rangstufen (als Ordnung) durch das ICTV.
- ↑ Lokis Schloss ist Namensgeber der Archaeengruppe Lokiarchaeota.
Einzelnachweise
- ↑ a b c d e f ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ Eugene V. Koonin, Natalya Yutin:Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism. In: Advances in Virus research, Band 103, AP 21. Januar 2019, doi:10.1016/bs.aivir.2018.09.002, S. 167–202. Die Klosneuviren sind teilweise als Klosneviruses fehlgeschrieben.
- ↑ Natalya Yutin, Philippe Colson, Didier Raoult, Eugene V. Koonin: Mimiviridae: clusters of orthologous genes, reconstruction of gene repertoire evolution and proposed expansion of the giant virus family. In: Virol J., 2013, 10, S. 106, 4. April 2013, doi:10.1186/1743-422X-10-106, PMC 3620924 (freier Volltext), PMID 23557328
- ↑ M. Suzan-Monti, B. La Scola, D. Raoult: Genomic and evolutionary aspects of Mimivirus. In: Virus Res. 117(1), 2006, S. 145–155.
- ↑ a b c List of the main “giant” viruses known as of today (March 2019). (PDF) Centre national de la recherche scientifique, Université Aix Marseille, März 2019.
- ↑ a b Morgan Gaia, Samia Benamar, Mondher Boughalmi, Isabelle Pagnier, Olivier Croce, Philippe Colson, Didier Raoult, Bernard La Scola: Zamilon, a Novel Virophage with Mimiviridae Host Specificity. In: PLoS One. Band 9(4), 2014, S. e94923, doi:10.1371/journal.pone.0094923, PMID 24747414, PMC 3991649 (freier Volltext). Online veröffentlicht am 18. April 2014
- ↑ a b c d e f Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses. In: Nature Communications, Band 9, Nr. 4881, 19. November 2018, doi:10.1038/s41467-018-07335-2.
- ↑ a b c d Jônatas Abrahão, Lorena Silva, Ludmila Santos Silva, Jacques Yaacoub Bou Khalil, Rodrigo Rodrigues, Thalita Arantes, Felipe Assis, Paulo Boratto, Miguel Andrade, Erna Geessien Kroon, Bergmann Ribeiro, Ivan Bergier, Herve Seligmann, Eric Ghigo, Philippe Colson, Anthony Levasseur, Guido Kroemer, Didier Raoult, Bernard La Scola: Tailed giant Tupanvirus possesses the most complete translational apparatus of the known virosphere. In: Nature Communications. Band 9, Nr. 1, 27. Februar 2018, doi:10.1038/s41467-018-03168-1 (Online).
- ↑ Daniel Lingenhöhl: Verschieben neue Riesenviren die Grenzen des Lebens? Spektrum.de, 28. Februar 2018
- ↑ Nadja Podbregar: Ungewöhnliche Riesenviren entdeckt. wissenschaft.de, 27. Februar 2018
- ↑ Riesenviren an der Grenze zum Leben, auf: scinexx.de, 28. Februar 2018
- ↑ a b David M. Needham, Susumu Yoshizawa, Toshiaki Hosaka, Camille Poirier, Chang Jae Choi, Elisabeth Hehenberger, Nicholas A. T. Irwin, Susanne Wilken, Cheuk-Man Yung, Charles Bachy, Rika Kurihara, Yu Nakajima, Keiichi Kojima, Tomomi Kimura-Someya, Guy Leonard, Rex R. Malmstrom, Daniel R. Mende, Daniel K. Olson, Yuki Sudo, Sebastian Sudek, Thomas A. Richards, Edward F. DeLong, Patrick J. Keeling, Alyson E. Santoro, Mikako Shirouzu, Wataru Iwasaki, Alexandra Z. Worden: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators, in: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424
- ↑ a b c d e f Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea… In: eLife Sciences, 7, März 2018, doi:10.7554/eLife.33014, ResearchGate
- ↑ V. M. Marcelino, M. V. P. C. Espinola, V. Serrano-Solis, S. T. Farias: Evolution of the genus Mimivirus based on translation protein homology and its implication in the tree of life. In: Genet.Mol.Res., 27. September 2017, 16(3), S. gmr16039784, doi:10.4238/gmr16039784
- ↑ Sharon C. Clouthier, E. Vanwalleghem, S. Copeland, C. Klassen, G. Hobbs, O. Nielsen, Eric D. Anderson: A new species of nucleo-cytoplasmic large DNA virus (NCLDV) associated with mortalities in Manitoba lake sturgeon Acipenser fulvescens. In: Dis Aquat Organ., 102(3), 28. Februar 2013, S. 195–209, doi:10.3354/dao02548, PMID 23446969
- ↑ Sharon C. Clouthier, E. Vanwalleghem, Eric D. Anderson: Sturgeon nucleo-cytoplasmic large DNA virus phylogeny and PCR tests. In: Dis Aquat Organ., 117(2), 9. Dezember 2015, S. 93–106, doi:10.3354/dao02937, PMID 26648102
- ↑ Sharon C. Clouthier, Eric D. Anderson, Gael Kurath, Rachel B. Breyta: Molecular systematics of sturgeon nucleocytoplasmic large DNA viruses. In: Mol Phylogenet Evol 128, Juli 2018, doi:10.1016/j.ympev.2018.07.019
- ↑ a b Jean-Michel Claverie, Chantal Abergel: Mimiviridae: An Expanding Family of Highly Diverse Large dsDNA Viruses Infecting a Wide Phylogenetic Range of Aquatic Eukaryotes. In: Viruses. 2018 Sep; 10(9), 18. September 2018, S. 506, doi:10.3390/v10090506, PMC 6163669 (freier Volltext), PMID 30231528
- ↑ a b Frederik Schulz, Simon Roux, David Paez-Espino, Sean Jungbluth, David A. Walsh, Vincent J. Denef, Katherine D. McMahon, Konstantinos T. Konstantinidis, Emiley A. Eloe-Fadrosh, Nikos C. Kyrpides, Tanja Woyke: Giant virus diversity and host interactions through global metagenomics. In: Nature Band 578, S. 432–436, 22. Januar 2020, doi:10.1038/s41586-020-1957-x, insbes. Fig. 1
- ↑ a b Frederik Schulz, Natalya Yutin, Natalia N. Ivanova, Davi R. Ortega, Tae Kwon Lee, Julia Vierheilig, Holger Daims, Matthias Horn, Michael Wagner: Giant viruses with an expanded complement of translation system components. In: Science. Band 356, Nr. 6333, 7. April 2017, ISSN 0036-8075, S. 82–85, doi:10.1126/science.aal4657, PMID 28386012 (englisch, Online).
- ↑ a b c Frederik Schulz, Natalya Yutin, Natalia N. Ivanova, Davi R. Ortega, Tae Kwon Lee, Julia Vierheilig, Holger Daims, Matthias Horn, Michael Wagner: Giant viruses with an expanded complement of translation system components. In: Science. Band 356, Nr. 6333, 7. April 2017, ISSN 0036-8075, S. 82–85, doi:10.1126/science.aal4657, PMID 28386012, bibcode:2017Sci...356...82S (englisch, Online)., UCPMS ID: 1889607, escholarship.org (PDF; 1,8 MB)
- ↑ a b c d e f g h Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere. In: Viruses, 11(4), März/April 2019, pii: E312, doi:10.3390/v11040312, PMC 6520786 (freier Volltext), PMID 30935049. Anm.: Die „Klosneuvirinae“ sind per Namensendung eine (vorgeschlagene) Unterfamilie der Mimiviridae, erst recht können die vorgeschlagenen Vertreter Yasminevirus und Fadolivirus nicht auch selbst neue Familien repräsentieren. Sie sind nicht die ersten isolierten Vertreter dieser Unterfamilie, das ist – wie an anderer stelle korrekt festgestellt wird, Bodo-saltans-Virus.
- ↑ NCBI: Bodo saltans virus (Spezies); abgerufen am 2. August 2019
- ↑ Hadjer Boudjemaa, Julien Andreani, Idir Bitam, Bernard La Scola: Diversity of Amoeba-Associated Giant Viruses Isolated in Algeria. In: Diversity, Band 12, Nr. 6, 215, 29. Mai 2020, doi:10.3390/d12060215.
- ↑ a b Leena Hussein Bajrai, Saïd Mougari, Julien Andreani, Emeline Baptiste, Jeremy Delerce, Didier Raoult, Esam Ibraheem Azhar, Bernard La Scola, Anthony Levasseur; Joanna L. Shisler (Hrsg.): Isolation of Yasminevirus, the First Member of Klosneuvirinae Isolated in Coculture with Vermamoeba vermiformis, Demonstrates an Extended Arsenal of Translational Apparatus Components, in: JVirol 94(1), Herbst 2019, doi:10.1128/JVI.01534-19
- ↑ Disa Bäckström, Natalya Yutin, Steffen L. Jørgensen, Jennah Dharamshi, Felix Homa, Katarzyna Zaremba-Niedwiedzka, Anja Spang, Yuri I. Wolf, Eugene V. Koonin, Thijs J. G. Ettema; Richard P. Novick (Hrsg.): Virus Genomes from Deep Sea Sediments Expand the Ocean Megavirome and Support Independent Origins of Viral Gigantism. ( des vom 17. April 2021 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. in: mBio Band 10, Nr. 2, 2019, doi:10.1128/mBio.02497-18
- ↑ a b NCBI: Mimivirus AB-566-O17 (Acronym: gvSAG AB-566-O17, gvSAG_O17) (Species)
- ↑ a b William H Wilson, Ilana C Gilg, Mohammad Moniruzzaman, Erin K Field, Sergey Koren, Gary R LeCleir, Joaquín Martínez Martínez, Nicole J Poulton, Brandon K Swan, Ramunas Stepanauskas, Steven W Wilhelm: Genomic exploration of individual giant ocean viruses. In: ISME Journal, 11(8), August 2017, S. 1736–1745, doi:10.1038/ismej.2017.61, PMC 5520044 (freier Volltext), PMID 28498373, nature, PDF (PDF; 746 kB) Associated Data (Supplements): Supplementary Dataset 1 (gvSAG AB-572-A11), Fig. 2 (gvSAG AB-566-O17)
- ↑ Vincent Racaniello, David Tuller, Gertrud U. Rey: Bodo saltans virus, an abundant giant aquatic Mimivirus. virology blog, 28. Dezember 2017
- ↑ NCBI: Satyrvirus (species)
- ↑ NCBI: Faunusvirus (species)
- ↑ NCBI: Gaeavirus (species)
- ↑ NCBI: Homavirus (species)
- ↑ NCBI: Barrevirus (species)
- ↑ NCBI: Dasosvirus (species)
- ↑ NCBI: Edafosvirus (species)
- ↑ NCBI: Terrestrivirus (species)
- ↑ NCBI: Harvfovirus (species)
- ↑ NCBI: Hyperionvirus (species)
- ↑ a b c d List of the main “giant” viruses known as of today. (PDF) Centre national de la recherche scientifique, Université Aix Marseille, 18. April 2018.
- ↑ a b David Paez-Espino, Jinglie Zhou, Simon Roux, Stephen Nayfach, Georgios A. Pavlopoulos, Frederik Schulz, Katherine D. McMahon, David Walsh, Tanja Woyke, Natalia N. Ivanova, Emiley A. Eloe-Fadrosh, Susannah G. Tringe, Nikos C. Kyrpides: Diversity, evolution, and classification of virophages uncovered through global metagenomics. In: Microbiome, Band 7, Nr. 157, 10. Dezember 2019, doi:10.1186/s40168-019-0768-5
- ↑ a b Weijia Zhang et al.: Four novel algal virus genomes discovered from Yellowstone Lake metagenomes. In: Scientific Reports, 2015, 5, Nr. 15131, doi:10.1038/srep15131, siehe Fig. 6
- ↑ a b Sebastian G. Gornik, Ian Hu, Imen Lassadi, Ross F. Waller: The Biochemistry and Evolution of the Dinoflagellate Nucleus. In: Microorganisms, 7(8), 28. Juni 2019, S. 245, doi:10.3390/microorganisms7080245.
- ↑ a b Shengzhong Xu, Liang Zhou, Xiaosha Liang, Yifan Zhou, Hao Chen, Shuling Yan, Yongjie Wang; Julie K. Pfeiffer (Hrsg.): Novel Cell-Virus-Virophage Tripartite Infection Systems Discovered in the Freshwater Lake Dishui Lake in Shanghai, China. ( des vom 11. April 2021 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. In: Journal of Virology, 18. Mai 2020, doi:10.1128/JVI.00149-20, PMID 32188734
- ↑ a b Jonathan Filée: Giant viruses and their mobile genetic elements: the molecular symbiosis hypothesis. In: Current Opinion in Virology, Band 33, Dezember 2018, S. 81–88; bioRxiv: 2018/04/11/299784 (Preprint-Volltext)
- ↑ Natalya Yutin et al.: Origin of giant viruses from smaller DNA viruses not from a fourth domain of cellular life. In: Virology, Volumes 466–467, Oktober 2014, S. 38–52, doi:10.1016/j.virol.2014.06.032
- ↑ ICTV: Virus Taxonomy. Abgerufen am 24. November 2022.
- ↑ P Colson, G Fournous, SM Diene, D Raoult: Codon usage, amino acid usage, transfer RNA and amino-acyl-tRNA synthetases in mimiviruses. In: Intervirology, 2013, 56(6), S. 364–375. doi:10.1159/000354557
- ↑ Bärbel Hauröder, Liane Junglas, Silke Loch, Rolf Michel, Karl-Dieter Müller, Claudia Wylezich: Experimental co-infection of Saccamoeba lacustris with Mimivirus-like Giant virus and a small Satellite virus, in: Open Agrar, 15. Mai 2018
- ↑ Yoshitoshi Ogura, Tetsuya Hayashi, Shoko Ueki: Complete Genome Sequence of a Phycodnavirus, Heterosigma akashiwo Virus Strain 53. In: Microbiology, September 2016, doi:10.1128/genomeA.01279-16, PMID 27834719, mra.asm.org (PDF)
- ↑ Fumito Maruyama, Shoko Ueki: Evolution and Phylogeny of Large DNA Viruses, Mimiviridae and Phycodnaviridae Including Newly Characterized Heterosigma akashiwo Virus. In: Front. Microbiol., 30. November 2016, doi:10.3389/fmicb.2016.01942, PMC 5127864 (freier Volltext), PMID 27965659
- ↑ Torill Vik Johannessen, Gunnar Bratbak, Aud Larsen, Hiroyuki Ogatac, Elianne S. Egged, Bente Edvardsen, Wenche Eikremd, Ruth-Anne Sandaa: Characterisation of three novel giant viruses reveals huge diversity among viruses infecting Prymnesiales (Haptophyta), in: Virology, Band 476, Februar 2015, S. 180–188, doi:10.1016/j.virol.2014.12.014, PMID 25546253, insbes. Fig. 4 und Fig. S2
- ↑ Lucie Gallot-Lavallée, Guillaume Blanc: A Glimpse of Nucleo-Cytoplasmic Large DNA Virus Biodiversity through the Eukaryotic Genomics Window. In: Viruses, 2017 Jan; 9(1), S. 17. PMC 5294986 (freier Volltext), PMID 28117696
- ↑ Autor=Nathan Zauberman, Y. Mutsafi, D. B. Halevy, E. Shimoni, E. Klein, C. Xiao, S. Sun, A. Minsky: Distinct DNA exit and packaging portals in the virus Acanthamoeba polyphaga mimivirus. In: PLoS Biol. Band 6, Nr. 5, 2008, S. e114, doi:10.1371/journal.pbio.0060114, PMID 18479185, PMC 2430901 (freier Volltext).
- ↑ M. G. Fischer, C. A. Suttle: A Virophage at the Origin of Large DNA Transposons. In: Science. Band 332, Nr. 6026, 2011, S. 231–234, doi:10.1126/science.1199412, PMID 21385722.
- ↑ Anthony Levasseur, Meriem Bekliz, Eric Chabrière, Pierre Pontarotti, Bernard La Scola, Didier Raoult: MIMIVIRE is a defence system in mimivirus that confers resistance to virophage. In: Nature, 2016, doi:10.1038/nature17146.
- ↑ Ewen Callaway: CRISPR-like ‘immune’ system discovered in giant virus. In: Nature:News, 29. Juni 2016
- ↑ Meriem Bekliz, Philippe Colson, Bernard La Scola: The Expanding Family of Virophages, in: MDPI Viruses 8(11), Special Issue Viruses of Protozoa, 23. November 2016, 317; doi:10.3390/v8110317
